Artículo original
Iber-Diaz P, Senén-Carramolino R, Palma-Fernández J C, Iglesias-Linares A. Reabsorción radicular apical externa tras la ortodoncia: factores clínicos y diagnósticos y análisis de redes de genes relacionados. Cient. Dent. 2022; 19; 2; 79-89
Reabsorción radicular apical externa tras la ortodoncia: Factores clínicos y diagnósticos y análisis de redes de genes relacionados
Introducción: La reabsorción radicular apical externa (EARR) provocada por las fuerzas ortodóncicas representa uno de los efectos iatrogénicos más indeseables del tratamiento.
Se pretende establecer una relación entre las variables diagnósticas y clínicas del tratamiento con la aparición y severidad de la reabsorción radicular apical externa agresiva (aEARR). Adicionalmente, la red de interrelaciones genéticas sirve para la generación de hipótesis en la correlación de variaciones genéticas, previamente asociadas con aEARR, con otras enfermedades.
Métodos: Se realizó un estudio de asociación mediante una selección de 240 pacientes clasificados en dos grupos, basándose en la presencia o ausencia de aEARR.
Se realizó un análisis descriptivo y una regresión logística binaria condicional tipo backward entre las variables y el desarrollo de aEARR.
Se seleccionaron 5 variantes (STAG2, RP1-30E17.2, P2RX7, SPP1 y TNFRSF11A) asociadas a una mayor predisposición al desarrollo de aEARR y se realizó un análisis de redes.
Resultados: Exclusivamente la variable tiempo de tratamiento obtuvo resultados estadísticamente significativos (IC: 95%; p=0,007) en relación con la aEARR.
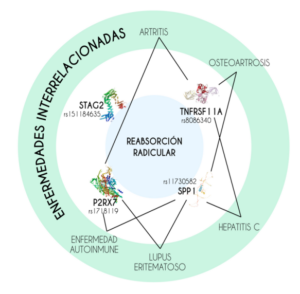
En el análisis de redes se encontró relación de 4 variantes genéticas con diferentes etapas del proceso patológico de la EARR que se relacionaron con 6 patologías: artritis, osteoartrosis, enfermedad autoinmune, lupus eritematoso, hepatitis C y EARR.
Conclusiones: El único factor que se asoció con una mayor prevalencia de aEARR es el tiempo de duración del tratamiento.
Cualquier vía que pueda tener relación con el proceso inflamatorio y, por tanto, con el proceso reabsortivo, puede tener una implicación mayor/menor de manera directa o indirecta en el desarrollo de la EARR.
Introduction: External apical root resorption (EARR) caused by orthodontic forces represents one of the most undesirable iatrogenic effects of treatment.
The aim of this study is to establish a relation between the diagnostic and clinical variables of treatment with the appearance and severity of aggressive external apical root resorption (aEARR). Additionally, the network of genetic interrelationships offers the generation of hypotheses in the correlation of genetic variations, previously associated with aEARR, with other diseases.
Methods: An association study was performed using a selection of 240 patients classified into two groups based on the presence or absence of aEARR. A descriptive analysis of the data along with a backward conditional binary logistic regression was performed between the variables and aEARR.
Five variants (STAG2, RP1-30E17.2, P2RX7, SPP1 and TNFRSF11A) associated with an increased predisposition to aEARR were selected and network analysis was performed.
Results: Only the treatment time variable obtained statistically significant results (CI: 95%; p=0.007) in relation to aEARR.
In the network analysis, 4 genetic variants were found to be related to different stages of the pathological process of aEARR, which were associated with 6 pathologies: arthritis, osteoarthrosis, autoimmune disease, lupus erythematosus, hepatitis C and EARR.
Conclusions: The only factor associated with a higher prevalence of aEARR is the treatment duration.
Any pathway that may be related to the inflammatory process and therefore to the resorptive process may have a greater/lesser involvement directly or indirectly in the development of aEARR.
La reabsorción radicular apical externa (EARR) provocada por las fuerzas ortodóncicas representa uno de los efectos iatrogénicos más indeseables del tratamiento, secundarios a la fuerza mecánica durante el movimiento ortodóncico, que provoca una pérdida irreversible de la estructura de la raíz del diente en el tercio apical. La EARR se manifiesta sobre todo en sus formas leves a moderadas; sin embargo, el tipo más agresivo (frecuencia < 1-5%; > 5 mm de pérdida apical) podría comprometer la viabilidad dentaria.
La EARR de cualquier grado de severidad representa un proceso patológico complejo con múltiples factores etiológicos de riesgo que, hasta la fecha, no han sido completamente determinados. Se han asociado varios factores diagnósticos y clínicos con la EARR. Factores como el tiempo de tratamiento y la cantidad de movimiento apical, se han vinculado con la EARR, pero estos hallazgos muestran cierto grado de inconsistencia y controversia en la literatura. Tanto es así que la aparición y gravedad de la EARR siguen siendo impredecibles y no se explican de un modo completo a través de variables exclusivamente de naturaleza clínica.
En este contexto, el componente genético y su contribución a este efecto iatrogénico es un tema de importancia crítica que está recibiendo una destacada atención por su potencial aplicación en el ámbito clínico. Hasta la fecha, un número limitado de estudios de asociación genética ha aportado resultados preliminares que sugieren que algunas variantes genéticas (SNPs) podrían ejercer una influencia positiva o negativa sobre la EARR, a nivel de los genes IL116,21-25, TNFRSF11B26, P2RX718,19, SPP120,24, STAG2 o TNFRSF11A16,27 e IRAK128 .
Durante el proceso en el que se produce la EARR, existe una fase destructiva mediada por macrófagos tipo M1 que liberan citoquinas proinflamatorias (IL6, IL1𝛼, IL1𝛽, RANKL, TNF𝛼, NO y INF𝛾) 21,29-34. Cuando el estímulo de fuerza mecánica ejercida por la aparatología ortodóncica desaparece, se inicia una fase de reparación, en la que predominan los macrófagos tipo M231,32,35. De esta manera, las alteraciones cualitativas y cuantitativas en los mediadores implicados en el movimiento dentario están determinadas por diversas variantes genéticas que pueden aumentar la susceptibilidad a sufrir EARR36.
A pesar de los estudios mencionados de EARR, apenas existe evidencia científica sobre la forma en que los factores genéticos podrían estar específicamente asociados con el fenotipo más grave de EARR, es decir, el EARR agresiva (aEARR). Por todo ello, esta investigación encuentra su justificación directa en tanto en cuanto los resultados de este estudio puedan suponer un avance en el conocimiento de los aspectos básicos de esta patología, con una capacidad de traslación a la práctica diaria del clínico. Sustentados en los avances informáticos y la gestión adecuada de las bases de datos en el ámbito de la genética, es posible establecer relaciones entre los polimorfismos y las vías proteínicas, y su vínculo con ciertas patologías. De esta forma, antes de comenzar con el tratamiento en un futuro sería deseable poder establecer el grado de susceptibilidad del paciente a sufrir reabsorción radicular y con ello valorar los beneficios y riesgos de un modo individualizado.
El presente estudio tiene como objetivo establecer una relación entre las variables diagnósticas y clínicas del tratamiento de ortodoncia con la aparición y severidad de la aEARR. Adicionalmente, la red de interrelaciones genéticas sirve de base para la generación de hipótesis en la correlación de variaciones genéticas, previamente asociadas con aEARR, con otras enfermedades.
Diseño y población de estudio
Se ha diseñado un estudio de asociación para la identificación de diversos factores clínicos, diagnósticos y de tratamiento, con un riesgo imputado respecto a la afectación de aEARR. Adicionalmente, en una segunda etapa se planteó un estudio generador de hipótesis a partir de las variantes genéticas relacionadas con la aEARR y otras patologías. A partir de una muestra de 650 pacientes, se realizó una selección de 240 pacientes que habían finalizado el tratamiento ortodóncico en la Universidad Complutense de Madrid.
Todos los sujetos seleccionados para su inclusión en el grupo de afectación severa (aEARR ≥ 5mm) o control (EARR< 5mm) se asignaron a través de selección radiológica, empleando medición radiográfica por duplicado y a doble ciego (P.I.D y R.S.C) sobre ortopantomografías y radiografías laterales de cráneo ya obtenidas y que se emplean para el diagnóstico y tratamiento de rutina en ortodoncia.
Todos los pacientes seleccionados cumplieron los criterios de inclusión previamente validados en la literatura37: tratamiento ortodóncico completo, sin alteraciones en los tejidos duros, sin graves malformaciones craneofaciales, sin traumas dentales, sin endodoncias en los dientes estudiados, formación radicular completa de incisivos superiores y duración del tratamiento menor de 5 años. El estudio se desarrolló con pleno conocimiento y consentimiento informado de cada sujeto, estando sometido a las directrices y principios éticos aplicables a la investigación médica en personas humanas, tal y como se expresa en la Declaración de Helsinki38 y con aprobación para la experimentación por parte del Comité de Ética Institucional [Código:17/038-E/2017].
Registros clínicos y diagnósticos
Los parámetros diagnósticos y clínicos registrados fueron los empleados en trabajos previos de referencia37. De modo específico se registraron 24 variables clínicas y 19 variables diagnósticas previamente asociadas en la literatura con influencia sobre la EARR de grado leve/medio11,39-41.
Método de medición radiográfica
Los sujetos se clasificaron y dividieron en dos grupos, basándose en la presencia o ausencia del fenotipo de aEARR. En las radiografías panorámicas, como variable dependiente de interés para cada sujeto, se determinó el incisivo superior con mayor grado de afectación en términos de reabsorción radicular apical externa después del tratamiento de ortodoncia. Se empleó metodología descrita por Linge y Linge42 y modificada por Brezniak y cols.43 para la medición digital de EARR EARR = TL1 – TL2 x (CL1/CL2), con el fin de establecer la diferencia entre el tamaño radicular al inicio y al final del tratamiento. El desplazamiento apical y la variación de la inclinación de los dientes se cuantificaron mediante la superposición de mediciones radiográficas en una radiografía lateral, utilizando una versión modificada del método descrito por Baccetti y cols.44.
Análisis de redes genéticas y de proteínas. Generación de hipótesis
Se analizaron cinco variantes genéticas asociadas a una mayor predisposición al desarrollo de aEARR con un grado de asociación estadística de p > 1×10-4 identificadas en un análisis genómico de 14717 variantes37. Los SNPs analizados fueron: STAG2, gen del antígeno estromal 2 (rs151184635) y RP1-30E17.2, gen Vega basado en clones (rs55839915) (ambos localizados en el cromosoma X), SPP1, osteopontina (rs11730582); P2RX7, receptor purinérgico 7 PX2 (rs1718119) y TNFRSF11A, miembro 11A de la superfamilia de receptor del TNFα (factor de necrosis tumoral alfa) (rs8086340). Estos SNPs se localizan en los cromosomas 4, 12 y 18, respectivamente.
Para establecer una asociación entre las variaciones genéticas que actúan a nivel del desarrollo de aEARR y de las relaciones que tienen a nivel proteínico molecular de vías patológicas y de enfermedades, se analizaron mediante Cytoscape 3.9.1, (The Cytoscape Consortium), se establecieron relaciones con GeneMANIA 3.5.2 (Genome Canada, through the Ontario Genomics Institute) y, de esta manera, fue posible generar nuevas hipótesis en la regulación e interrelación de los genes y sus posibles implicaciones en otros procesos patológicos. A través de STRING 11.5 (https://string-db.org/), se estudiaron las relaciones de las proteínas señaladas con otras proteínas y su implicación en diversas patologías. Se exploró The Genotype-Tissue Expression (GTEx) project; neXtProt; UniProt; GeneCards Suite, en el que están incluidos: GeneCards®: The Human Gene Database, MalaCards (https://www.malacards.org): The human disease database y PathCards: Human Biological pathway unification y de Novo Nordisk foundation Center for Protein Research (DISEASES) (https://diseases.jensenlab.org/Search).
Análisis estadístico
Error y precisión del método
Todas las mediciones descritas se realizaron por un examinador previamente entrenado y calibrado. Asimismo, un segundo examinador experimentado replicó las mediciones en un subconjunto de 50 pacientes. Adicionalmente, en términos categóricos se cuantificó el valor Kappa marginal; se realizó una comparación estadística de mediciones duplicadas a doble ciego realizadas en 20 pacientes elegidos aleatoriamente y con una separación en el tiempo de 20 días de intervalo. Para los cálculos se utilizó la t de Student; también se calculó el coeficiente de correlación intraclase (ICC) para determinar el grado de concordancia en términos absolutos tanto para los errores intra como interobservador. La precisión de la medición se obtuvo a partir de la ecuación: SE = √(Σd2/2n).
Análisis descriptivo y de asociación
Se realizó un análisis descriptivo de las variables cuantitativas y categóricas basadas en factores de diagnóstico o tratamiento (media, desviación estándar -SD-, rangos, frecuencias y distribuciones). Además, se realizó una regresión logística binaria condicional tipo backward entre todas las variables y el desarrollo de aEARR. Se calculó la odds ratio (OR) con un intervalo de confianza del 95% y una significación estadística con un valor de p<0,05 [SPSS versión 27.0. Lead Technologies, Chicago, IL, USA].
La reproducibilidad y concordancia de las mediciones obtuvieron resultados satisfactorios (k:1,00; IC: 0,93) y un error del método de 0,04 mm.
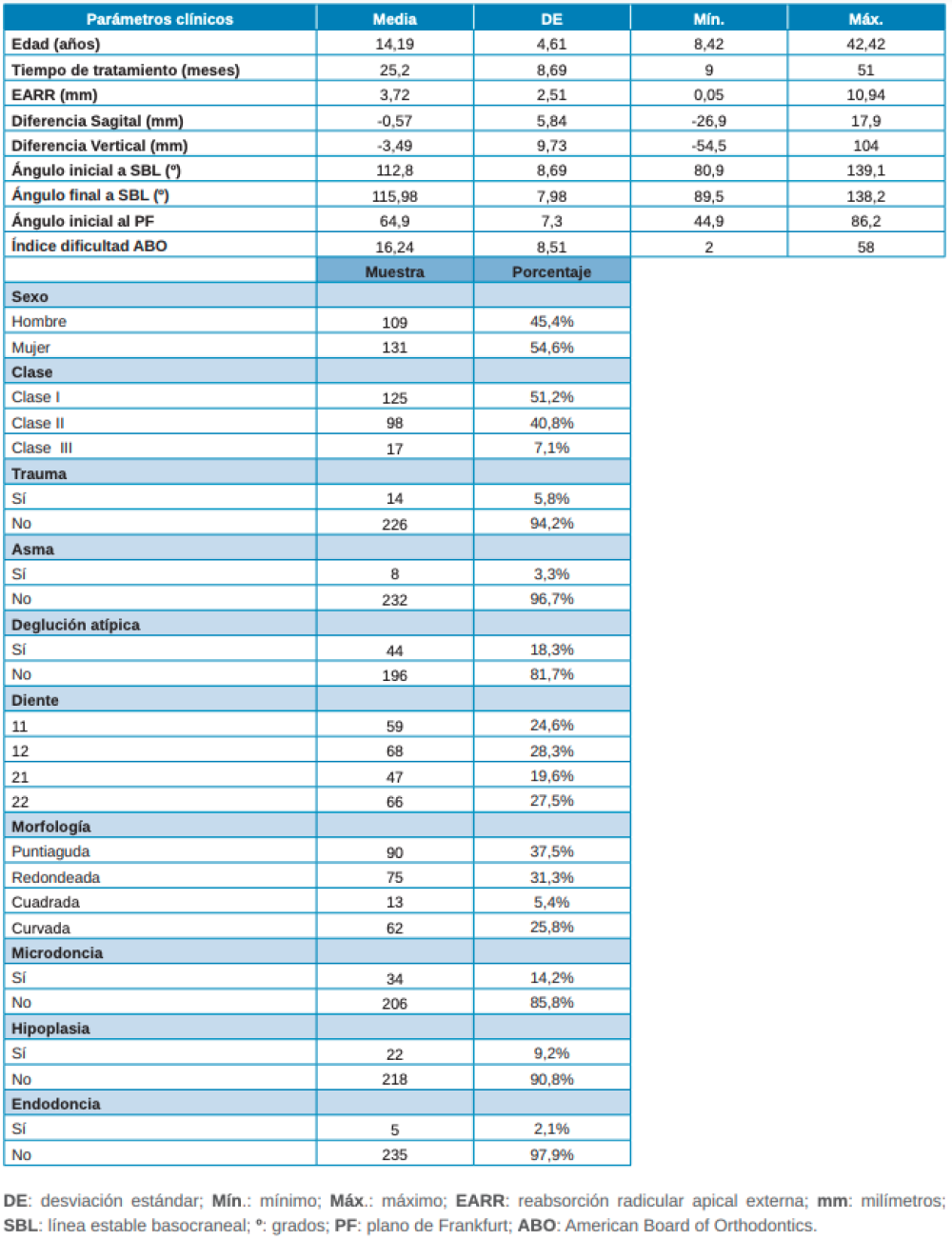
La muestra se compuso de 109 hombres y 131 mujeres, con una media de edad de 14,19 ± 4,61 años, un tiempo medio de tratamiento de ortodoncia de 25,2 ± 8,69 meses y una EARR media de 3,72 ± 2,51 mm. Los resultados del análisis descriptivo se pueden consultar en la Tabla 1.
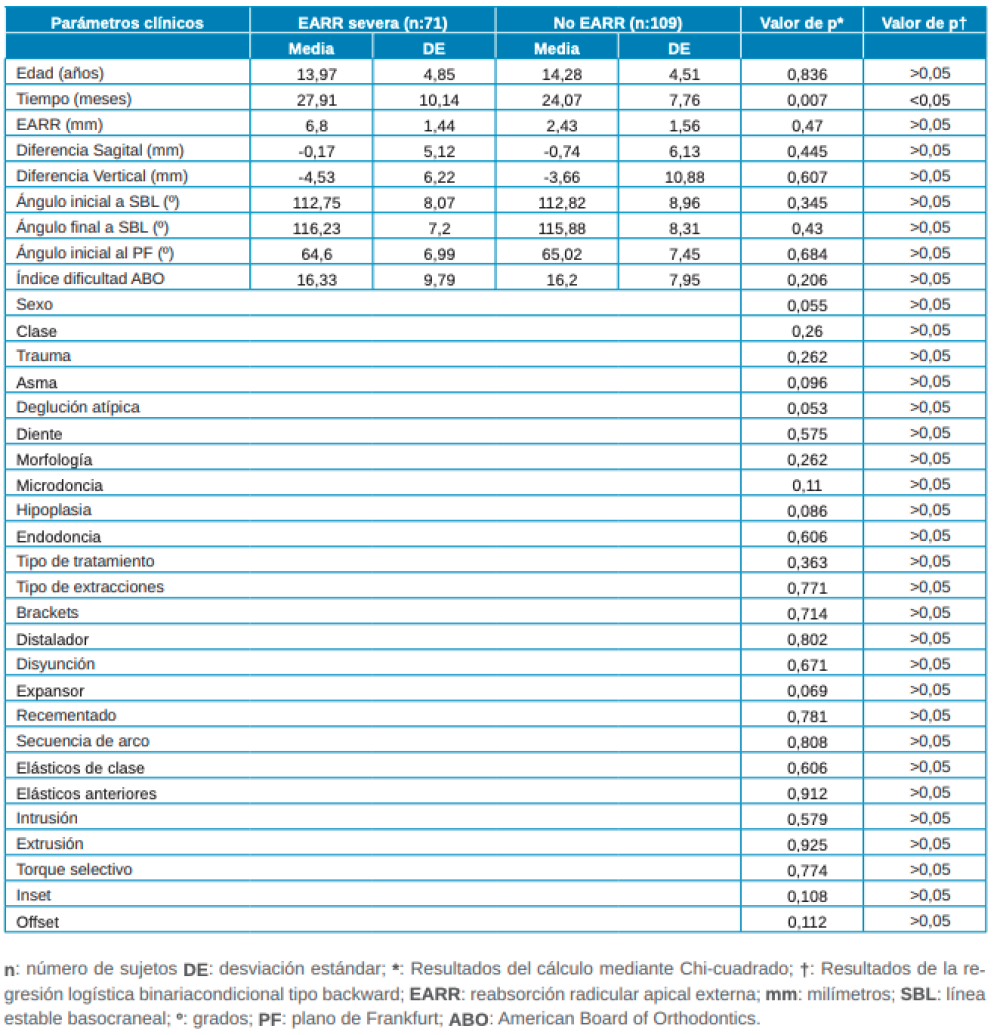
Tras realizar un análisis del riesgo de aEARR y su potencial asociación con las características clínicas, se observó que exclusivamente la variable tiempo de tratamiento obtuvo resultados estadísticamente significativos (IC: 95%; p=0,007) en relación con la aparición de aEARR. Los resultados completos derivados de del análisis de regresión logística se pueden consultar en la Tabla 2
muestra según la EARR.
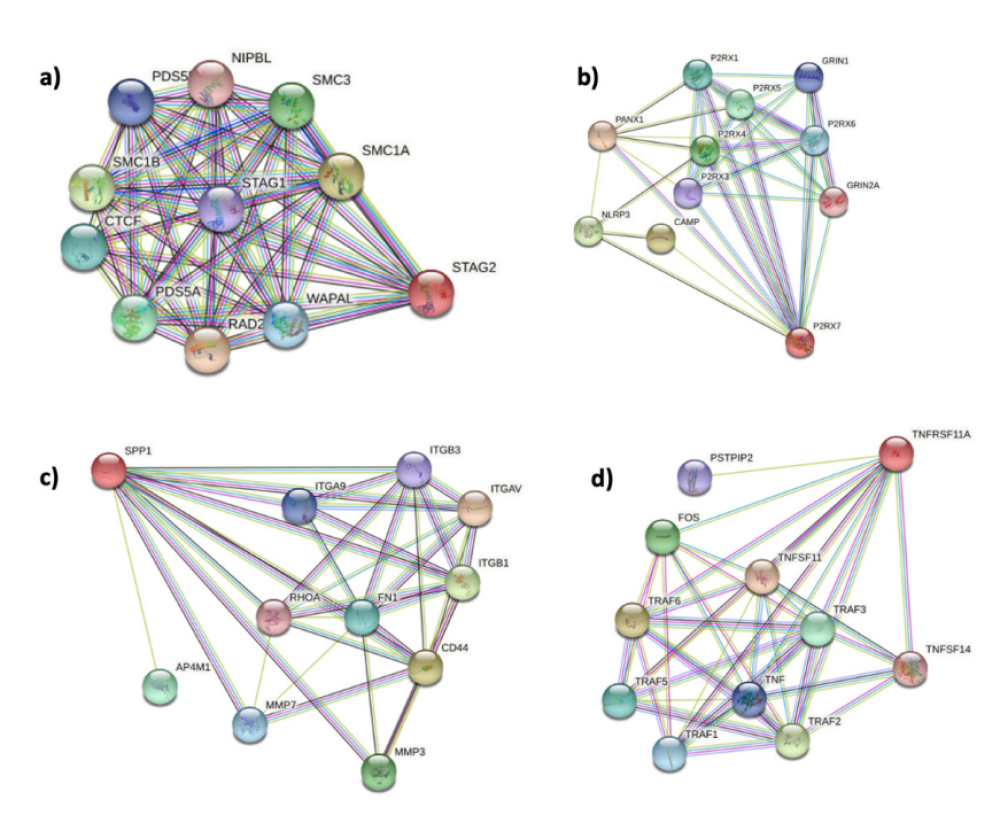
A partir de cinco variantes asociadas a una mayor predisposición al desarrollo de aEARR, de entre un total de 14717 variantes analizadas (STAG2, RP1-30E17.2, P2RX7, SPP1 y TNFRSF11A), se realizó un análisis de redes genéticas e interrelaciones. En la Figura 1 se describen las diferentes interrelaciones que presentan las proteínas específicas de los genes en los que se localizan las variantes identificadas en asociación con la reabsorción radicular. En la Figura 2 se describe de modo específico la relación de cada una de las proteínas con los procesos biológicos en los que se ven implicadas. De esta forma, el gen STAG2 codifica para la proteína STAG2 la subunidad de cohesina SA-2 (antígeno estromal), que es necesaria para la cohesión de las cromátidas hermanas después de la replicación del ADN y actúa de manera sustancial en la división celular y en la organización celular45. El gen que codifica para P2RX7 (receptor de purinas 7 P2X), es un receptor de ATP que actúa como canal iónico activado por ligando (ATP). Es responsable de la lisis de macrófagos dependiente de ATP y funciona como un receptor depurador en el reconocimiento y absorción de células apoptóticas. El gen SPP1 codifica para la proteína SPP1 (Fosfoproteína 1 secretada u osteopontina), ayuda a la reestructuración de la matriz mineralizada y actúa como una citocina. Asimismo, está implicada en la respuesta inflamatoria, la regulación de la transcripción de ADN y la adhesión celular. La proteína TNFRSF11A, miembro 11A de la superfamilia del receptor del factor de necrosis tumoral (receptor para TNFSF11/RANKL/TRANCE/OPGL), es esencial para la osteoclastogénesis mediada por RANKL. Participa en la regulación de las interacciones entre las células T y las células dendríticas y moléculas de células dendríticas. Actúa también en las mismas vías moleculares de la inflamación y de la reabsorción. No se han encontrado relaciones del gen basado en clones (Vega) RP1-30E17.2 con otros genes ni proteínas o patologías en los sistemas investigados, exceptuando la EARR.
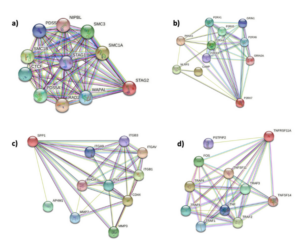
b) P2RX7. c) SPP1. d) TNFRSF11A. Imágenes obtenidas de
Protein-Protein Interaction Networks Functional Enrichment
Analysis (https://string-db.org/).

Adicionalmente, se relacionaron estas 5 proteínas conjuntamente a través de STRING. En una primera búsqueda simple, no se observó relación directa entre ellas, por lo que se realizó un análisis de cluster en el que se agruparon las proteínas en función de su actividad biológica y se obtuvieron 3 cluster (Figura 3). El primer cluster hace referencia a STAG2 y otras proteínas que se consideran implicadas en la cohesión de las cromátidas hermanas y en el proceso de división celular y nuclear. En el cluster 2 se encontraron implicadas P2RX7 y TNFRSF11A, involucradas en la regulación positiva de la secreción de prostaglandinas, regulación de la reabsorción ósea y respuesta a lipopolisacáridos. En el tercer cluster se encuentra la SPP1, asociada al complejo de contención proteínica (protein-containing complex), que son conjuntos estables de dos o más macromoléculas.
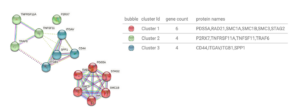
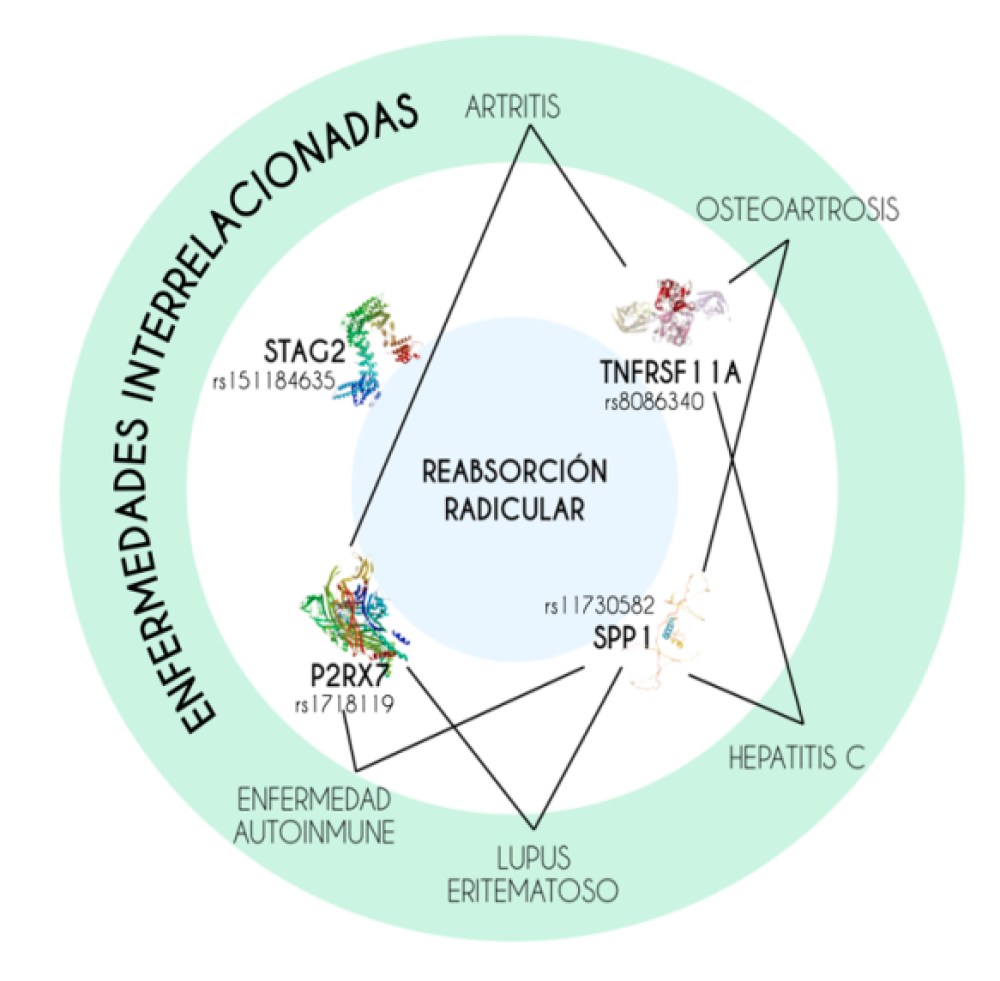
Se realizó un análisis cruzado de búsqueda, a partir de los genes seleccionados, en la base de datos DISEASE. El gen SPP1 se relacionó con 322 enfermedades diferentes; P2RX7 con 165 patologías; TNFRSF11A con 130 enfermedades, STAG2 con 65 enfermedades y, por último, el gen basado en clones Vega (RP1- 30E17.2) únicamente se relacionó con la reabsorción radicular. De modo más específico, en el análisis de relación de las variantes genéticas específicas identificadas se observó una relación de todas ellas con la reabsorción radicular, así como una asociación de la artritis (P2RX7: rs1718119 y TNFRSF11A: rs8086340), osteoartrosis y hepatitis C (SPP1: rs11730582 y TNFRSF11A: rs8086340), lupus eritematoso y enfermedad autoinmune (SPP1: rs11730582 y P2RX7: rs1718119). Estas implicaciones en las enfermedades señaladas se resumen en la Figura 4.
variantes específicas analizadas de cada gen identificado en el
estudio de Iber-Díaz de 2020. (https://www.malacards.org).
El estudio de la genética de cualquier enfermedad trata de identificar y localizar las variantes que puedan estar asociadas a una mayor o menor severidad y/o predisposición al desarrollo de dicha patología46,47. Además de los genes implicados, existen factores clínicos, diagnósticos y de tratamiento que pueden afectar a su aparición.
Los resultados del presente estudio sugieren que el tiempo de tratamiento desempeña un papel significativo en el desarrollo de la aEARR. Otros autores9,30,48-50, sin embargo, no observan esta relación y esto puede deberse a la diferencia en la metodología de cuantificación, origen, tipo y número de muestra, así como a la interacción de otros factores de confusión que puedan limitar la interpretación de las relaciones que puedan existir. Los tratamientos de larga duración suelen implicar modificaciones sobre las mecánicas y tipos de fuerzas ortodóncicas habituales9,18,24,35,51-59, pero, igualmente, es importante tener en cuenta también la predisposición individual al desarrollo de EARR.
Existe evidencia científica de la relación de factores genéticos que afectan al desarrollo de la EARR durante el tratamiento de ortodoncia16,17,21,24,52,60. Los polimorfismos genéticos pueden afectar o incluso modular la predisposición de la EARR asociada al tratamiento ortodóncico (estímulo mecánico). Las modificaciones genéticas pueden darse en las bases del ADN de una región que codifique para una proteína que influya en el proceso de la EARR. Esa modificación puede inducir un cambio en la respuesta habitual al mismo estímulo19. Por ello, es importante analizar la vía patológica de la reabsorción radicular para entender su relación con los genes analizados en este estudio.
El proceso biológico por el que se produce la reabsorción radicular durante el tratamiento de ortodoncia se inicia con la tensión mecánica prolongada sobre el ligamento periodontal. A pesar de su complejidad, se ha asociado a un incremento de la cantidad de macrófagos tipo M1, que aumenta la producción de citoquinas proinflamatorias como son IL6, I-1α, IL-1β y IL6 y también RANKL32,41. En esta vía, el gen TNFRSF11A codifica para el receptor RANK, cuyo papel es determinante en la recepción de la señal citoquínica del estímulo pro-reabsortivo, así como en la actividad del receptor de señalización transmembrana61 y la actividad del receptor activado por el factor de necrosis tumoral62. La unión de TNF con su receptor facilita la actividad osteoclástica y producción celular, por lo que modificaciones genéticas en este punto pueden suponer una alteración respecto a la severidad de la reabsorción.
La unión del osteoclasto a la superficie mineralizada requiere la expresión de integrinas, como la osteopontina (OPN). El gen SPP1 codifica para la OPN y está implicado en la adhesión celular y en la respuesta inflamatoria como la diferenciación de osteoblastos y la regulación positiva de la reabsorción ósea63-65, lo que relaciona esta modificación genética con el proceso de EARR en la capacidad de adhesión de las células clásticas.
Una vez que el osteoclasto está unido a la superficie por medio de las integrinas, éste empieza a crear una laguna en la superficie calcificada por medio de la liberación de moléculas (Cl- , MMP, H+) que producen la exposición del cemento radicular y, de esta forma, se inicia la EARR. En este punto es plausible la interacción del producto proteico codificado por el gen P2RX7, ya que expresa el receptor celular de la ATP y aumenta la liberación de H+ y además está involucrado en la regulación negativa de: reabsorción ósea, mineralización ósea66, respuesta a la isquemia67 y producción de IL-1 β (la citoquina más proinflamatoria), lo que pudiese mediar en un aumento de la capacidad reabsortiva y aumento del proceso de la reabsorción.
La variante rs151184635 del gen STAG2, a nivel biológico está implicada en la división celular, el ciclo de la célula meiótica, la cohesión de las cromátidas hermanas68 y el ensamblaje del huso mitótico69,70. Es posible que pueda relacionarse con alteraciones en la proliferación de células clásticas y, por tanto, con la división celular de los macrófagos. Las células clásticas son partícipes del desarrollo de la reabsorción radicular, por lo que alteraciones en la expresión de estas proteínas están directamente relacionadas con la proliferación y actividad de estas. Además, sabemos que tienen expresión en células linfocitarias, así como en fibroblastos, lo que hace que esta relación sea mucho más estrecha con la patología que nos ocupa71. El gen RP1-30E17.2 (rs55839915) ha sido relacionado únicamente en un estudio con sólo una patología: la EARR37, esto puede deberse a su escasa caracterización por el momento en las bases de datos.
En términos generales, el aumento o sobreproducción de IFN-γ, IL-1α, IL-1β, IL-18, TNFα, IL-2, IL-6 y factor estimulador de colonias de macrófagos (M-CSF), puede estar asociado con un aumento en la posibilidad de desarrollar aEARR. Esto se debe a que la EARR está basada en la actividad de los macrófagos72.
Atendiendo a la relación de los genes con diversas enfermedades, más de 680 enfermedades se han visto asociadas con alguno de los genes. En el que se ha visto una implicación con un mayor número de enfermedades es el SPP1, con más de 320, mientras que para el que menos se han identificado es para el gen STAG2, con 65; posiblemente por ser de más reciente interés en la comunidad científica. De todas estas enfermedades, solo 19 de ellas se han conseguido relacionar con los 4 genes a la vez. De estas, la alergia, el síndrome linfoproliferativo y la gripe son las que mayor relación tienen con los procesos por los que se da la reabsorción radicular, el proceso de inflamación y reabsorción. De esta forma, vías moleculares que están implicadas en la reabsorción, se ven relacionadas también en estas patologías.
De las enfermedades relacionadas con las alteraciones específicas (SNPs) de los genes seleccionados, encontramos: artritis73, enfermedad autoinmune74, osteoartritis75, lupus eritematoso76 y hepatitis C77, en las que hay anomalías en la respuesta inflamatoria. Aunque se ha visto que estas enfermedades comparten vías de actuación, se deben considerar para futuras investigaciones las posibles conexiones que existen en los pacientes cuyas alteraciones hemos analizado.
De los resultados de la investigación se puede concluir, respecto a los factores clínicos, diagnósticos y de tratamiento, que el único factor que se asoció con una mayor prevalencia de aEARR en la presente muestra es el tiempo de duración del tratamiento. El resto de las variables de estas categorías no se vieron relacionadas directamente con el desarrollo de la patología.
En un análisis exhaustivo de las relaciones de los genes elegidos, sus implicaciones moleculares, así como la influencia de estos en diversas enfermedades, cualquier vía que pueda tener relación con el proceso inflamatorio y, por tanto, con el proceso reabsortivo, puede tener una implicación mayor/menor de manera directa o indirecta en el desarrollo de la EARR. El estudio de estas interacciones, tanto a nivel molecular, funcional como de enfermedades, es importante para desarrollar nuevas hipótesis que puedan investigarse en un futuro y que ayuden a esclarecer el inicio, desarrollo y severidad de la EARR.
Al Ilustre Colegio Oficial de Odontólogos de la I Región por promover la investigación y hacer posible este proyecto gracias a la beca de Investigación concedida.
Sondeijker CFW, Lamberts AA, Beckmann SH y cols. Development of a clinical practice guideline for orthodontically induced external apical root resorption. Eur J Orthod. 2020;42(2):115-24.
Ahuja R, Almuzian M, Khan A, Pascovici D, Dalci O, Darendeliler MA. A preliminary investigation of short-term cytokine expression in gingival crevicular fluid secondary to high-level orthodontic forces and the associated root resorption: case series analytical study. Prog Orthod. 2017;18(1):23.
Fontana ML, de Souza CM, Bernardino JF y cols. Association analysis of clinical aspects and vitamin D receptor gene polymorphism with external apical root resorption in orthodontic patients. Am J Orthod Dentofacial Orthop. 2012;142(3):339-47.
Al-Qawasmi RA, Hartsfield JK, Jr., Everett ET y cols. Genetic predisposition to external apical root resorption. Am J Orthod Dentofacial Orthop. 2003;123(3):242-52.
Harris EF, Kineret SE, Tolley EA. A heritable component for external apical root resorption in patients treated orthodontically. Am J Orthod Dentofacial Orthop. 1997;111(3):301-9.
Matsuyama Y, Tsakos G, Listl S, Aida J, Watt RG. Impact of Dental Diseases on Quality-Adjusted Life Expectancy in US Adults. J Dent Res. 2019;98(5):510-6.
Artun J, Van ‘t Hullenaar R, Doppel D, Kuijpers-Jagtman AM. Identification of orthodontic patients at risk of severe apical root resorption. Am J Orthod Dentofacial Orthop. 2009;135(4):448-55.
Mirabella AD, Artun J. Risk factors for apical root resorption of maxillary anterior teeth in adult orthodontic patients. Am J Orthod Dentofacial Orthop. 1995;108(1):48-55.
Currell SD, Liaw A, Blackmore Grant PD, Esterman A, Nimmo A. Orthodontic mechanotherapies and their influence on external root resorption: A systematic review. Am J Orthod Dentofacial Orthop. 2019;155(3):313-29.
Guo Y, He S, Gu T, Liu Y, Chen S. Genetic and clinical risk factors of root resorption associated with orthodontic treatment. Am J Orthod Dentofacial Orthop. 2016;150(2):283-9.
Segal GR, Schiffman PH, Tuncay OC. Meta analysis of the treatment-related factors of external apical root resorption. Orthod Craniofac Res 2004;7:71-8.
Mohandesan H, Ravanmehr H, Valaei N. A radiographic analysis of external apical root resorption of maxillary incisors during active orthodontic treatment. Eur J Orthod. 2007;29(2):134-9.
Aman C, Azevedo B, Bednar E, y cols. Apical root resorption during orthodontic treatment with clear aligners: A retrospective study using cone-beam computed tomography. Am J Orthod Dentofacial Orthop. 2018;153(6):842-51.
Nowrin SA, Jaafar S, Ab Rahman N, Basri R, Alam MK, Shahid F. Association between genetic polymorphisms and external apical root resorption: A systematic review and meta-analysis. Korean J Orthod. 2018;48(6):395-404.
Tarallo F, Chimenti C, Paiella G, Cordaro M, Tepedino M. Biomarkers in the gingival crevicular fluid used to detect root resorption in patients undergoing orthodontic treatment: A systematic review. Orthod Craniofac Res. 2019;22(4):236-47.
Al-Qawasmi RA, Hartsfield JK, Everett ET y cols. Genetic predisposition to external apical root resorption in orthodontic patients: Linkage of chromosome-18 marker. J. Dent. Res. 2003;82:356-360.
Borges de Castilhos B, Machado de Souza C, Simas Netta Fontana MLS, Pereira FA, Tanaka OM, Trevilatto PC. Association of clinical variables and polymorphisms in RANKL, RANK, and OPG genes with external apical root resorption. Am J Orthod Dentofacial Orthop. 2019;155(4):529-42.
Pereira S, Lavado N, Nogueira L, Lopez M, Abreu J, Silva H. Polymorphisms of genes encoding P2X7R, IL-1B, OPG and RANK in orthodontic-induced apical root resorption. Oral Dis. 2014;20(7):659-67.
Sharab LY, Morford LA, Dempsey J y cols. Genetic and treatment-related risk factors associated with external apical root resorption (EARR) concurrent with orthodontia. Orthod Craniofac Res. 2015;18 Suppl 1:71-82.
Iglesias-Linares A, Yanez-Vico RM, Moreno-Fernandez AM, MendozaMendoza A, Orce-Romero A, SolanoReina E. Osteopontin gene SNPs (rs9138, rs11730582) mediate susceptibility to external root resorption in orthodontic patients. Oral Dis. 2014;20(3):307-12.
Iglesias-Linares A, Yanez-Vico RM, Ortiz-Ariza E y cols. Postorthodontic external root resorption in root-filled teeth is influenced by interleukin-1beta polymorphism. J Endod. 2012;38(3):283- 7.
Bastos HN, Antao MR, Silva SN y cols. Association of polymorphisms in genes of the homologous recombination DNA repair pathway and thyroid cancer risk. Thyroid. 2009;19(10):1067-75.
Gulden N, Eggermann T, Zerres K, Beer M, Meinelt A, Diedrich P. Interleukin-1 polymorphisms in relation to external apical root resorption (EARR). J Orofac Orthop. 2009;70(1):20-38.
Linhartova P, Cernochova P, Izakovicova Holla L. IL1 gene polymorphisms in relation to external apical root resorption concurrent with orthodontia. Oral Dis. 2013;19(3):262-70.
Iglesias-Linares A, Yanez-Vico RM, Ballesta S y cols. Interleukin 1 gene cluster SNPs (rs1800587, rs1143634) influences post-orthodontic root resorption in endodontic and their contralateral vital control teeth differently. Int Endod J. 2012;45(11):1018-26.
Shank S, Shank K, Caudill R, Foroud T, Wetherill L, Weaver M. Evaluation of SNPs in orthodontic patients with root resorption. J Dent Res.2007;86:1042-49.
Chung PY, Beyens G, Riches PL y cols. Genetic variation in the TNFRSF11A gene encoding RANK is associated with susceptibility to Paget’s disease of bone. J Bone Miner Res. 2010;25(12):2592- 605.
Pereira S, Nogueira L, Canova F, Lopez M, Silva HC. IRAK1 variant is protective for orthodontic-induced external apical root resorption. Oral Dis. 2016;22(7):658- 64.
Consolaro A, Bianco DA. Tooth resorptions are not hereditary. Dental Press J Orthod. 2017;22(4):22-7.
Darcey J, Qualtrough A. Resorption: part 1. Pathology, classification and aetiology. BrDent J. 2013;214(9):439-51.
He D, Kou X, Yang R y cols., M1-like Macrophage Polarization Promotes Orthodontic Tooth Movement. J Dent Res. 2015;94(9):1286-94.
He D, Kou X, Luo Q y cols. Enhanced M1/M2 macrophage ratio promotes orthodontic root resorption. J Dent Res 2015;94(1):129-39.
Tyrovola JB, Spyropoulos MN, Makou M, Perrea D. Root resorption and the OPG/ RANKL/RANK system: a mini review. J Oral Sci. 2008;50(4):367-76.
Tyrovola JB, Perrea D, Halazonetis DJ, Dontas I, Vlachos IS, Makou M. Relation of soluble RANKL and osteoprotegerin levels in blood and gingivalcrevicular fluid to the degree of root resorption after orthodontic tooth movement. J Oral Sci. 2010;52(2):299-311.
Iglesias-Linares A, Morford LA, Hartsfield JK. Bone Density and Dental External Apical Root Resorption. Curr Osteoporos Rep. 2016;14(6):292-309.
Fernández R, Tobón D, Osorno K, Zuluaga OE. Polimorfismo genetico para la interleukina-1 beta como modulador de los procesos de reabsorción ósea. Revisión de Literatura. Rev. CES Odont. 2012;25(1) 92-101.
Iber-Díaz P, Senén-Carramolino R, Iglesias-Linares A, Fernández-Navarro P, Flores-Mir C, Yañez-Vico RM. GWAS of Post-Orthodontic Aggressive External Apical Root Resorption Identified Multiple Putative Loci at X-Y Chromosomes. J Pers Med. 2020;10(4):169.
World Medical A. World Medical Association Declaration of Helsinki: ethical principles for medical research involving human subjects. JAMA. 2013;310(20):2191-4.
Roscoe MG, Meira JB, Cattaneo PM. Association of orthodontic force system and root resorption: A systematic review. Am J Orthod Dentofacial Orthop. 2015;147(5):610-26.
Çelikoglu M, Halıcıoğlu K, Çağlaroğlu M. Association between root resorption incident to orthodontic treatment and treatment factors. Eur J Orthod 2013;35:273.
Iglesias-Linares A, Hartsfield JK Jr. Cellular and Molecular Pathways Leading to External Root Resorption. J Dent Res. 2017;96:145-152.
Linge L, Linge BO. Patient characteristics and treatment variables associated with apical root resorption during orthodontic treatment. Am J Orthod Dentofacial Orthop. 1991;99(1):35-43.
Brezniak N, Goren S, Zoizner, y cols. The use of an individual jig in measuring tooth length changes. Angle Orthod. 2004;74(6):780-5.
Baccetti T, McGill JS, Franchi L, McNamara JA Jr, Tollaro I. Skeletal effects of early treatment of Class III malocclusion with maxillary expansion and face-mask therapy. Am J Orthod Dentofacial Orthop. 1998;113(3):333-43.
Ren H, Chen J, Deng F, Zheng L, Liu X, Dong Y. Comparison of cone-beam computed tomography and periapical radiography for detecting simulated apical root resorption. Angle Orthod. 2013;83(2):189-195.
Al-Harazi O, Al Insaif S, Al-Ajlan MA, Kaya N, Dzimiri N, Colak D. Integrated Genomic and Network-Based Analyses of Complex Diseases and Human Disease Network. J Genet Genomics. 2016;43(6):349-67.
Boyle EA, Li YI, Pritchard JK. An Expanded View of Complex Traits: From Polygenic to Omnigenic Cell. 2017;169(7):1177-86.
Weltman B, Vig KW, Fields HW, Shanker S, Kaizar EE. Root resorption associated with orthodontic tooth movement: a systematic review. Am J Orthod Dentofacial Orthop. 2010;137(4):462-76.
Darcey J, Qualtrough A. Resorption: part 2. Diagnosis and management. Br Dent J. 2013;214(10):493-509.
Motokawa M, Sasamoto T, Kaku M y cols. Association between root resorption incident to orthodontic treatment and treatment factors. Eur J Orthod. 2012;34(3):350-6.
Maués CP, do Nascimento RR, Vilella Ode V. Severe root resorption resulting from orthodontic treatment: prevalence and risk factors. Dental Press J Orthod. 2015;20(1):52-8.
Hartsfield JK, Everett, Al-Qawasmi RA. Genetic factors in external apical root resorption and orthodontic treatment. Crit Rev Oral Biol Med. 2004;15(2):115-22.
Fernandes LQP, Figueiredo NC, Montalvany Antonucci CC, Lages EMB, Andrade IJr, Capelli J. Predisposing factors for external apical root resorption associated with orthodontic treatment. Korean J. Orthod. 2019,49: 310–8.
Tsichlaki A, Chin SY, Pandis N, Fleming PS. How long does treatment with fixed orthodontic appliances last? A systematic review. Am. J. Orthod. Dentofac. Orthop. 2016;149:308-18.
Jiang RP, McDonald JP, Fu MK. Root resorption before and after orthodontic treatment: A clinical study of contributory factors. Eur. J. Orthod. 2010,32:693-7.
Kaklamanos EG, Makrygiannakis MA, Athanasiou AE. Does medication administration affect the rate of orthodontic tooth movement and root resorption development in humans? A systematic review. Eur. J. Orthod. 2020;42:407-14.
Zymperdikas VF, Yavropoulou MP, Kaklamanos EG, Papadopoulos MA. Effects of systematic bisphosphonate use in patients under orthodontic treatment: A systematic review. Eur. J. Orthod. 2020;42:60-71.
Sirisoontorn I, Hotokezaka H, Hashimoto M y cols. Tooth movement and root resorption; the effect of ovariectomy on orthodontic force application in rats. Angle Orthod. 2011;81:570-7.
Wang J, Rousso C, Christensen BI, Li P y cols. Ethnic differences in the root to crown ratios of the permanent dentition. Orthod. Craniofac. Res. 2019;22:99-104.
Zhou J, Hu H, Huang R. A pilot study of the metabolomic profiles of saliva from female orthodontic patientswith external apical root resorption. Clin Chim Acta. 2018;478:188-93.
Anderson DM. A homologue of the TNF receptor and its ligand enhance T-cell growth and dendritic-cell function. Nature. 1997;390:175-9.
Hanada R. Central control of fever and female body temperature by RANKL/RANK. Nature, 2009;462:505-9.
Gaudet P, Livstone MS, Lewis SE, Thomas PD. Phylogenetic-based propagation of functional annotations within the Gene Ontology consortium. Brief Bioinform. 2011;12:449-62.
Klaning E. Multiple low-affinity interactions support binding of human osteopontin to integrin alphaXbeta2. Biochim Biophys Acta. 2015;1854:930-38.
Leitner L. Osteopontin promotes aromatase expression and estradiol production in human adipocytes. Breast Cancer Res Treat. 2015;154:63-9.
Ousingsawat J. Anoctamin 6 mediates effects essential for innate immunity downstream of P2X7 receptors in macrophages. Nat Commun. 2015;6:6245.
Cavaliere F. Up-regulation of P2X2, P2X4 receptor and ischemic cell death: prevention by P2 antagonists. Neuroscience 2003;120:85-98.
Gregson HC, Schmiesing JA, Kim J.- S, Kobayashi T, Zhou S, Yokomori K.A potential role for human cohesin in mitotic spindle aster assembly. J Biol Chem. 2001;276:47575-82.
Soardi FC, Machado-Silva A, Linhares ND y cols. Familial STAG2 germline mutation defines a new human cohesinopathy. NPJ Genom Med. 2017;2:7.
Mullegama SV. De novo loss-of-function variants in STAG2 are associated with developmental delay, microcephaly, and congenital anomalies. Am J Med Genet A. 2017;173:1319-27.
De Koninck. Funciones únicas de la cohesina-SA2 en proliferación y regulación génica Impacto sobre el desarrollo embrionario. En la Universidad Autónoma de Madrid:España;2020
Grom AA, Horne A, De Benedetti F. Macrophage activation syndrome in the era of biologic therapy. Nat Rev Rheumatol. 2016;12:259–68.
Saradna A, Do DC, Kumar S, Fu QL, Gao P. Macrophage polarization and allergic asthma. Transl Res. 2018;191:1-14.
Rose NR, Kong Y-CM, Sundick RS. The genetic lesions of autoimmunity. Clin. Exp. Immun. 1980;39:545-50.
Martel-Pelletier J, Barr AJ, Cicuttini FM, Conaghan PG, Cooper C, Goldring MB, Goldring SR, Jones G, Teichtahl AJ, Pelletier JP. Osteoarthritis. Nat Rev Dis Primers. 2016;13;2:16072.
Zucchi D, Elefante E, Calabresi E, Signorini V, Bortoluzzi A, Tani C. One year in review 2019: systemic lupus erythematosus. Clin Exp Rheumatol. 2019;37(5):715-22.
Chan ST, Ou JJ. Hepatitis C Virus-Induced Autophagy and Host Innate Immune Response. Viruses. 2017;9(8):224

Iber-Diaz, Paula
Estudiante doctorado en Ciencias Odontológicas en la Universidad Complutense de Madrid (UCM). Profesora Ayudante UCM. Máster de ortodoncia UCM.
Senén-Carramolino, Raquel
Estudiante doctorado en Ciencias Odontológicas en la UCM. Máster de ortodoncia UCM.
Palma-Fernández, Juan Carlos
Departamento Especialidades Clínicas Odontológicas UCM.
Iglesias-Linares, Alejandro
Departamento Especialidades Clínicas Odontológicas UCM.